Un nouveau niveau de régulation découvert dans la sporulation bactérienne
L’équipe Galinier et ses collaborateurs ont mis en évidence un mécanisme inédit de régulation de la sporulation chez Bacillus subtilis. En utilisant pour la première fois la méthode OOPS (Orthogonal Organic Phase Separation) sur une bactérie Gram positive, ils ont pu dresser une carte complète des protéines liant l’ARN (RBPome) au cours de la formation de spores.
Leurs analyses révèlent un remodelage majeur du RBPome pendant la sporulation, avec l’identification de plus de 1 500 protéines interagissant avec l’ARN, dont certaines jusque-là jamais associées à ce type de régulation. Parmi elles, des protéines métaboliques et des facteurs peu caractérisés comme KhpA et SpoVR semblent jouer un rôle clé dans l’adaptation à des conditions hostiles.
Ces découvertes ouvrent la voie à une meilleure compréhension de la différenciation bactérienne et apportent de nouvelles perspectives pour la lutte contre les pathogènes sporulants, avec des retombées potentielles en santé publique, sécurité alimentaire et biotechnologies.

Figure : Caractérisation du RBPome de Bacillus subtilis pendant la sporulation
A : Suivi en microscopie de Bacillus subtilis pendant le processus de sporulation. B : Présentation du protocole du OOPS. Pour purifier le RBPome impliqué dans la sporulation, nous avons réalisé des expériences de OOPS à 3, 7 et 12 h en milieu de sporulation (DSM). Les protéines libres (FP) et les RBP sont analysées (LC-MS) et quantifiées (LFQ). C : Filtres utilisés pour l’analyse des données protéomiques. Après utilisation de filtres de qualité, sur les 2731 protéines répertoriées initialement, il reste 1520 RBP.
A new layer of regulation uncovered in bacterial sporulation
Galinier team and its coll. have identified a previously unknown regulatory mechanism in the sporulation of Bacillus subtilis. For the first time, they applied the OOPS (Orthogonal Organic Phase Separation) approach to a Gram-positive bacterium, allowing a global mapping of RNA-binding proteins (the RBPome) during spore formation.
The study reveals a dramatic remodeling of the RBPome throughout sporulation, identifying over 1,500 RNA-binding proteins, including many not previously linked to RNA regulation. Remarkably, metabolic proteins and poorly characterised factors such as KhpA and SpoVR were found to interact with RNA, suggesting unexpected roles in bacterial adaptation to harsh environments.
These findings provide a new framework for understanding bacterial differentiation and survival, with potential repercussions for public health, food safety, and industrial applications.
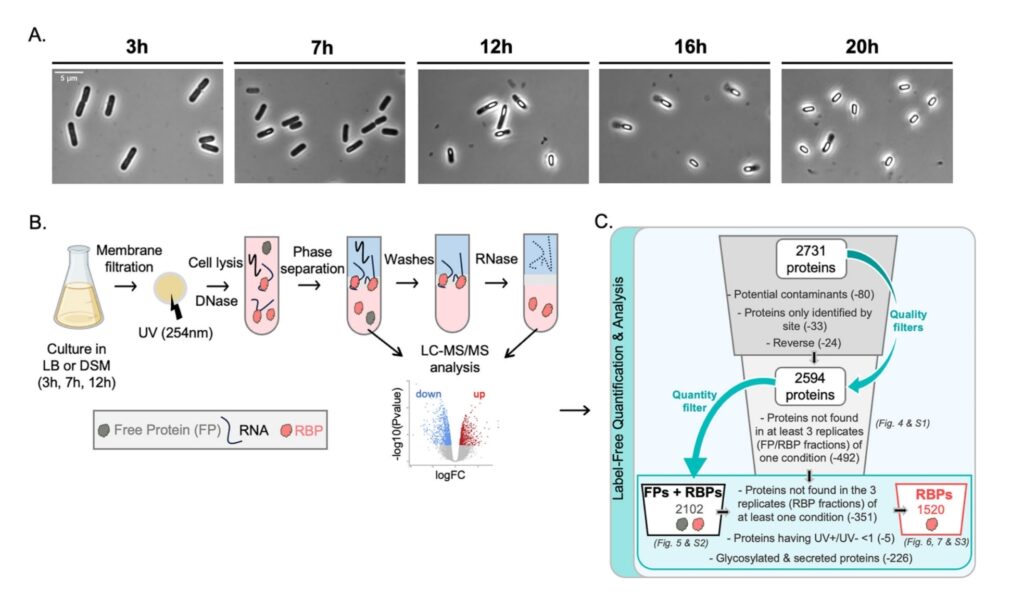
Figure: Characterization of the RBPome of Bacillus subtilis during sporulation
A: Microscopy monitoring of Bacillus subtilis during the sporulation process.
B: Overview of the OOPS protocol. To purify the RBPome involved in sporulation, OOPS experiments were performed at 3, 7, and 12 hours in sporulation medium (DSM). Free proteins (FP) and RNA-binding proteins (RBPs) were analyzed (LC-MS) and quantified (LFQ).
C: Filters used for proteomic data analysis. After applying quality filters, out of the initially identified 2,731 proteins, 1,520 RBPs remained.



